Sistemas polimórficos de interesse antropológico, médico e forense
1. Definição de polimorfismo
O genoma humano possui cerca de 3 bilhões de pares de bases (pb), embora essa quantidade varia de um indivíduo para outro. Entre dois genomas haploides existe cerca de 0,1% de variação que pode ser classificada em tipos, que veremos à continuação. O conjunto de alelos (formas alternativas) de uma sequência de DNA em uma população constitui o pool gênico da mesma. Este funciona como um reservatório de diversidade genética que pode servir como material para adaptação a mudanças no ambiente, por exemplo.
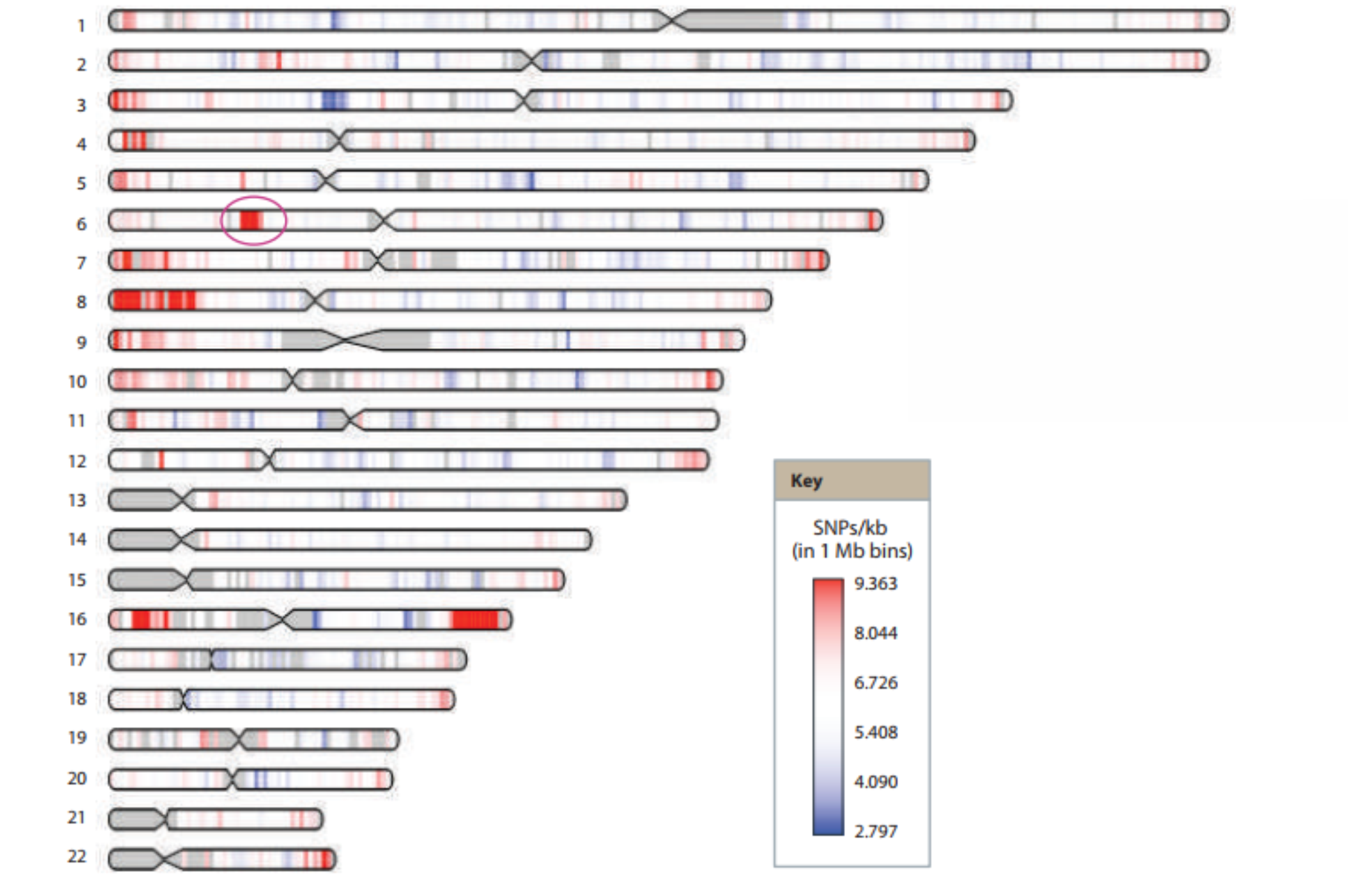
Dizemos que uma sequência é polimórfica quando a mesma apresenta, no mínimo, dois alelos na população, cada um com frequência >= 1%. Sequências polimórficas podem ser bialélicas (Ex.: inserção/deleção, substituições com dois alelos) ou multialélicas (Ex.: polimorfismos de número de cópias). O contrário de polimorfismo é o monomorfismo (ou sequência monoalélica) onde não ocorre variação. O conceito de polimorfismo é qualitativo, podendo uma sequência ser ou não ser polimórfica. Assim, o que pode variar entre distintas sequências é o seu grau de polimorfismo. Exemplo: como se observa na figura o loco HLA (do inglês Human Leucocite Antigen, antígeno leucocitário humano) destacado com um círculo vermelho, representa uma das regiões com maior densidade de polimorfismos de nucleotídeo único, ou SNPs. Dizemos que este loco possui um alto grau de polimorfismo.
É importante diferenciar o conceito de variante. O termo variante pode ser utilizado para falar de um polimorfismo, mas o primeiro também abrange diferenças menos frequentes entre cópias do genoma cujos alelos não necessariamente atingem a frequência necessária para ser incluído na categoria de polimorfismo. Ex.: variantes podem ser singletons (um dos alelos foi detectado apenas uma vez na população) ou doubletons.
Por último o termo mutação indica uma mudança de novo (que ocorre pela primeira vez). No momento que ela ocorre existe como singleton. O resultado dos processos evolutivos fará com que essa mutação siga um dentre vários caminhos, desde aumentar a sua frequência e se tornar um polimorfismo até ser eliminada da população.
O que é necessário para que uma mutação se torne um polimorfismo?
Mutações são o resultado de danos ao DNA e falhas no sistema de reparo. A diversidade em um dado locus (ou local no genoma, em plural loci, em português loco) depende tanto da sua taxa de mutação como da seleção natural e da história populacional. Para que uma mutação se torne um polimorfismo esta deve:
- Ocorrer na linhagem germinativa (células que dão origem aos gametos).
- Permitir a sobrevivência e não afetar a reprodução. Ex.: Mudanças no número de cromossomos (aneuploidias) decorrem de falhas na segregação cromossômica na meiose por isso não são herdáveis.
- Cada alelo deve alcançar uma frequência >= 1% na população.
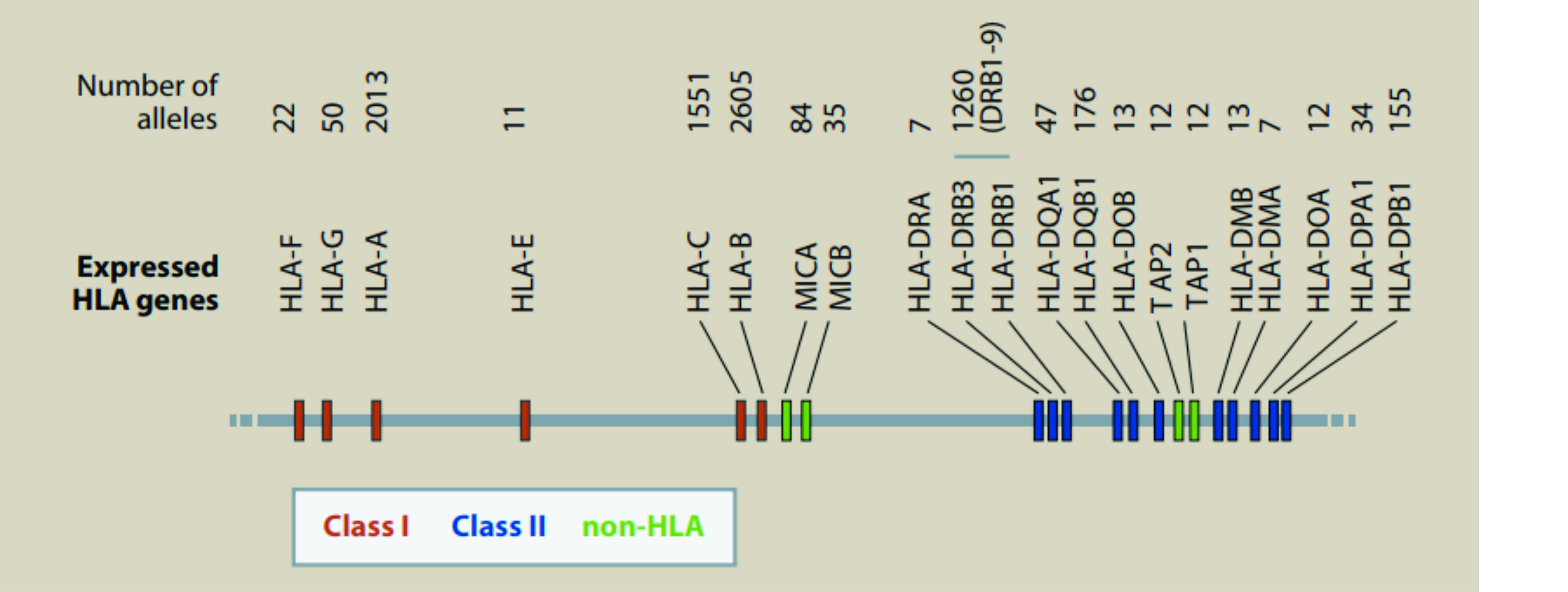
Os genes HLA apresentam antígenos aos linfócitos T, estando envolvidos com a resposta imune adaptativa. Diferentes alelos de cada gene individual variam na habilidade de apresentar antígenos de diferentes patógenos. O locus HLA (3,6 Mb no cromossomo 6) está dividido em 3 regiões, chamadas "classes". Nas regiões de classe I e II, antigos eventos de duplicação geraram distintos genes expressos e pseudogenes.
Os diferentes alelos são mantidos na população por seleção balanceadora, permitindo que a mesma esteja preparada para a sobrevivência a patógenos emergentes. Hoje são os principais determinantes da histocompatibilidade.
Tipos de marcadores genéticos
A começos do século XX, após a redescoberta dos trabalhos de Gregor Mendel (1822-1884) sobre hibridização de plantas, começa um período de extensa pesquisa no campo da genética de populações. Porém, durante essa primeira etapa os pesquisadores ainda não tinham as ferramentas para estudar diretamente os polimorfismos genéticos em humanos. Os primeiros trabalhos da área são voltados à observação indireta da variação genética, isto é, no nível das proteínas (produtos do DNA). Esse primeiro grupo de marcadores estudados são hoje chamados de Marcadores Clássicos e foram a base das primeiras descobertas em poliformismos.
Marcadores Clássicos
No grupo de marcadores clássicos podemos diferenciar dois subgrupos em função da forma de estudo dos mesmos:
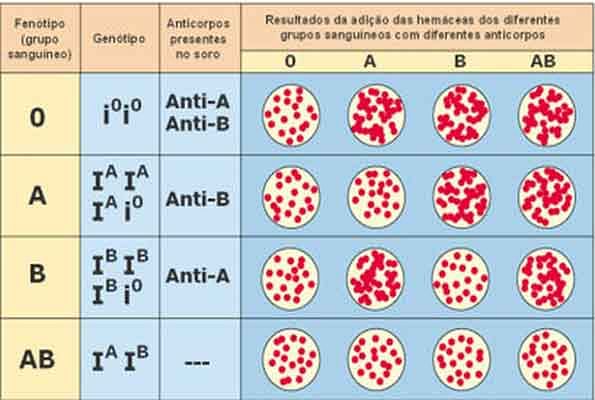
Marcadores imunogenéticos: a detecção da variação é baseada no reconhecimento de um antígeno por um anticorpo. Neste caso é o antígeno quem indica a variação no nível do DNA. O estudo pioneiro neste campo foi o realizado pelo casal polonês Hanka e Ludwik Hirszfeld, na Sérbia, durante a Primeira Guerra Mundial. Eles estudaram os grupos sanguíneos ABO em 8000 pessoas de diferentes nacionalidades. Outros estudos relevantes da época foram voltados para alelos HLA e para imunoglobulinas.
Marcadores bioquímicos: a variação genética, refletida em mudanças na cadeia de aminoácidos, é detectada através do padrão de migração da proteína em um gel de eletroforese. Isto se deve a que mudanças na cadeia de aminoácidos afetam a carga elétrica e a conformação da proteína, o que afeta a sua velocidade de migração no gel. Porém, essa abordagem apenas é útil quando a proteína a ser estudada se encontra em alta concentração, permitindo a sua visualização direta no gel, o que não é o caso de muitas proteínas humanas. Essa limitação foi contornada pelo uso de outro sistema de detecção, a tinção histoquímica. Neste caso, a proteína estudada deve ser uma enzima e sua presença no gel é detectada por um produto de reação de fácil visualização, como compostos coloreados. Desta forma, as mudanças no padrão de migração de proteínas em baixa concentração podem ser visualizadas.
Marcadores de DNA
A detecção direta de variações na sequência de nucleótidos do DNA começa a ser possível graças à descoberta, em 1970, das enzimas de restrição. A possibilidade de cortar e colar fragmentos de DNA da origem aos primeiros marcadores de DNA estudados: os polimorfismos de comprimento de fragmentos de restrição (ou RFLP, do inglês Restriction Fragment Lenght Polymorphism). Estes marcadores baseiam-se no reconhecimento pela enzima de restrição de sequências específicas de nucleótidos e na produção, após o corte, de fragmentos com comprimentos diferentes. A visualização dos fragmentos é realizada através da técnica de Southern blotting: o produto de reação enzimática é corrido em um gel de agarose e posteriormente transferido, por capilaridade, a uma membrana de nitrocelulose. Essa membrana é incubada com sondas de DNA marcado com nucleótidos radioativos, os quais são detectados pela exposição a raios-X. Fragmentos de DNA de comprimento conhecido, e também marcados radioativamente, são incluídos na corrida eletroforética inicial, permitindo estimar o tamanho dos fragmentos de restrição.