Completion requirements
View
Conteúdo do módulo
3. Sistemas uni e biparentais
Uma questão importantíssima que determina a aplicabilidade dos distintos polimorfismos, na prática, é a sua forma de herança. A mesma depende do cromossomo em que aquele polimorfismo está inserido.
Podemos dividir em:
Marcadores biparentais: autossomos, cromossomo X.
Marcadores uniparentais: cromossomo Y, DNAmt.
Marcadores biparentais
- Autossomos: 99,9% do DNA da célula.
- São os marcadores mais estudados para entender as bases genéticas de fenótipos.
- O seu estudo mostra a história completa de miscigenação de um indivíduo/população.
- Sofrem recombinação.
Marcadores Uniparentais
- DNAmt, cromossomo Y.
- Não sofrem recombinação, a única fonte de diversidade é a mutação.
- Uteis para conhecer linhagens.
- O seu estudo mostra apenas a história parcial de miscigenação de um indivíduo/população.
- Permitem detectar vieses sexuais nas contribuições ancestrais de uma população e outros fenômenos demográficos Ex.: patri ou matrilocalidade (em antropologia, descreve o comportamento das populações, se os casais tendem a morar na população de origem do individuo masculino ou femenino, respectivamente).
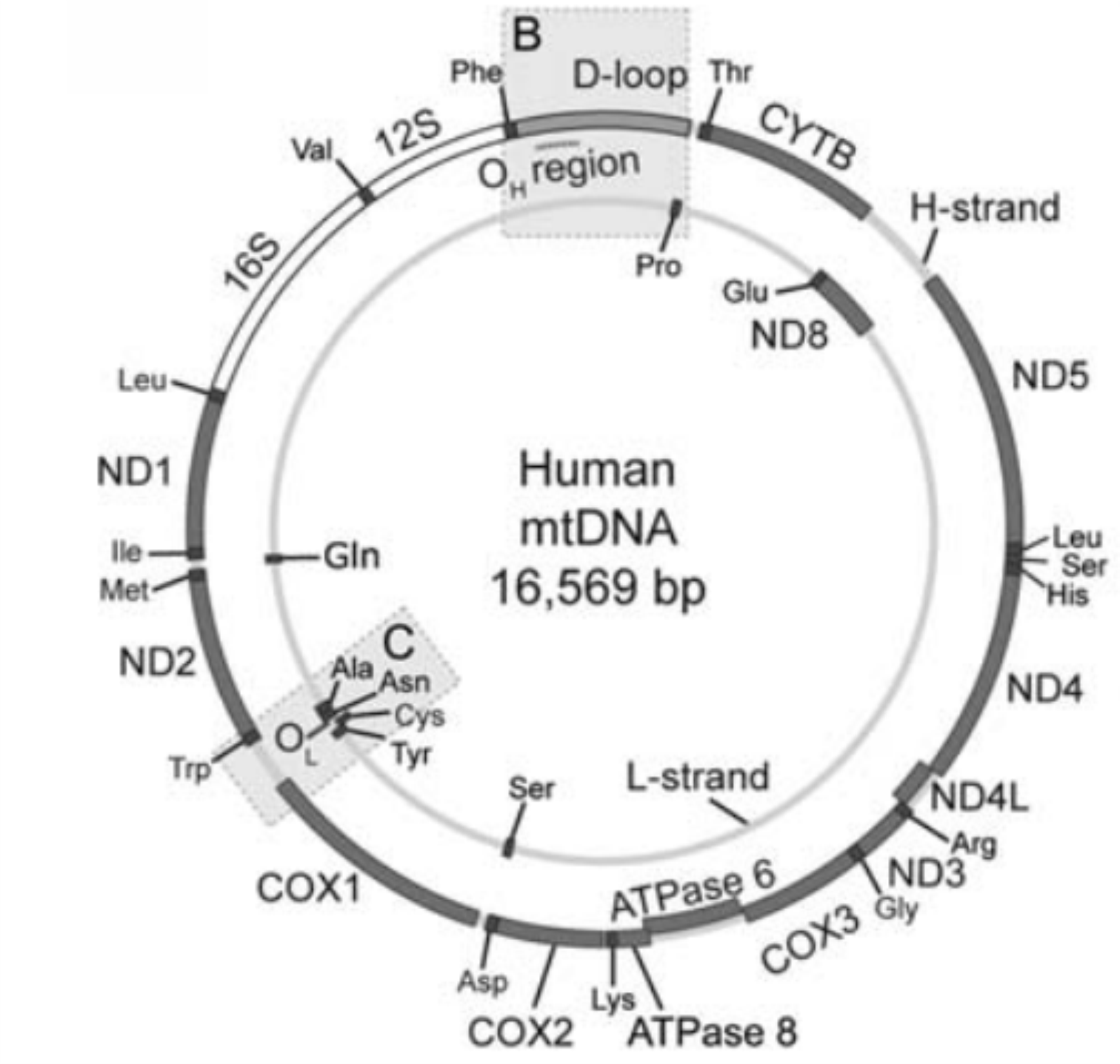
O DNAmt
O genoma mitocondrial tem sido amplamente utilizado no estudo de populações humanas devido a suas características:
- Ele é um genoma haploide pequeno, 16.569 pb.
- Genoma circular, compacto e sem íntrons (DNA não codificador é apenas 7%).
- Não possui histonas.
- Codifica apenas 37 genes:
- 13 envolvidos na fosforilação oxidativa.
- 2 RNAr
- 22 RNAt.
- Código genético diferente do nuclear.
- Alto número de cópias (cada célula somática possui centos de milhares de cópias).
- Herança materna.
- Marcadores estudados: SNPs e indels, sequenciamento completo e/ou apenas da região de controle. Essa região contem os promotores de transcrição para os RNAm policistrônicos.
- Velocidade de mutação é 10 X maior do que a do genoma nuclear. É possível estimar essa velocidade diretamente desde pedigrís.
- Velocidade de mutação varia entre as distintas partes do DNAmt, sendo 10 X maior nas regiões hipervariáveis I e II (HVSI e HVSII) na região de controle. Isto devido a:
- Envolvimento da mitocôndria na fosforilação oxidativa que produz radicais livres mutagênicos.
- Maior taxa de replicação que o DNA nuclear.
- Em cada célula há uma coexistência de cópias wild type (de tipo selvagem) e mutadas = heteroplasmia (o oposto é homoplasmia). Porém, o indivíduo apresenta um genoma mitocondrial predominante (o mais comum).
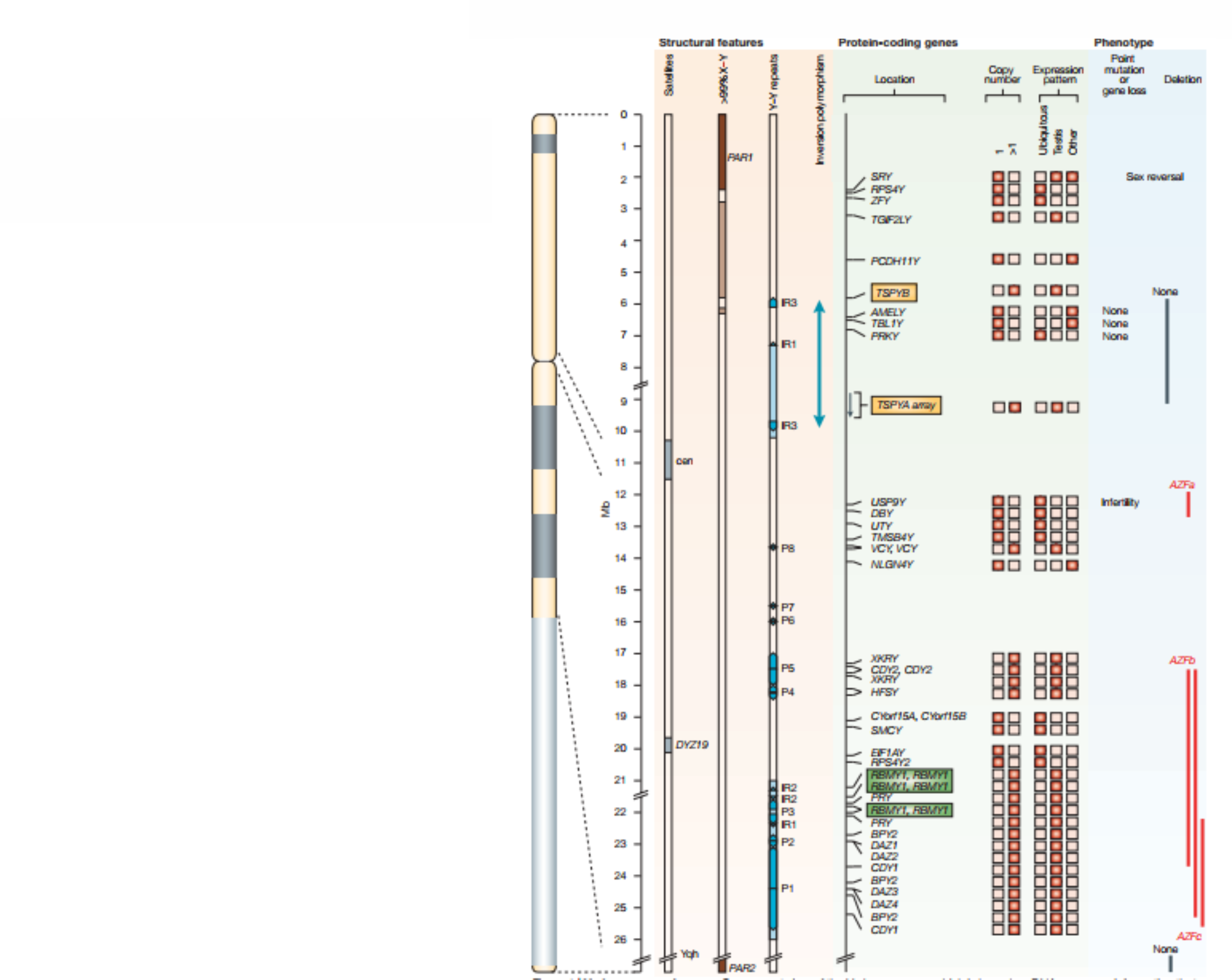
O cromossomo Y
- Genoma haploide de 60 Mb (2% do genoma humano haploide).
- ~95 % DNA não recombinante (NRY = non-recombining Y), haplótipos são passados intactos de pai para filho.
- Única fonte de variação é a mutação.
- ~5% é constituído pelas regiões pseudoautossômicas (PAR1 e PAR2, teloméricas) as quais recombinam com regiões homólogas no cromossomo X.
- > 50% de DNA não codificante.
- Tendência a degenerar: ao longo da sua evolução foi perdendo genes.
- ~200 genes, ~55 ativos principalmente envolvidos com desenvolvimento sexual e fertilidade masculina. Alguns genes possuem homólogos no cromossomo X.
- Inclui o gene SRY, determinante do sexo.
- Marcadores estudados: STRs, Alu, indels, SNPs, sequenciamento completo.